
Отчет по результатам генетического анализа образцов тканей мумий, найденных в Перу. Данный отчёт подготовлен в ноябре 2018 года.
Исполнители

- CEN4GEN labs (6756 – 75 Street NW Edmonton, AB Canada T6E 6T9) – подготовка и секвенирование образцов.
- ABRAXAS BIOSYSTEMS S.A.P.I. DE C.V. (Мексика) – компьютерный анализ данных.
После предварительного анализа на качество из 7 представленных образцов были отобраны 3 образца для последующего анализа.
Образцы для анализа
| Обозначение | Оригинальное название | Условное имя | Рисунок |
| Ancient-0002 | Neck Bone Med Seated 00-12 Victoria 4 | Виктория | Рис.3.117 |
| Ancient-0003 | 1 Hand 001 | Отдельная рука с 3 пальцами | Рис.3.118 |
| Ancient-0004 | Momia 5 — DNA | Виктория | Рис.3.117 |
Для этих образцов были выполнены следующие операции:
- Извлечение ДНК.
- Проверка качества ДНК.
- Мультиплицирование ДНК.
- Создание библиотеки ДНК.
- Секвенирование ДНК.
- Формирование очищенных секвенированных данных.
- Контроль качества.
- Предварительных анализ наложением ДНК ридов на геном человека.
- Анализ для выделения коротких ридов ДНК типичных для древних ДНК.
- Наложение ДНК ридов Ancient0003 на имеющиеся библиотеки генома человека.
- Митохондриальный анализ для обнаружения вариантов D-петель и других информативных участков для определения митохондриальных гаплотипов.
- Определение половой принадлежности образцов Ancient0003.
- Выявление возможных посторонних организмов в образцах.
- Анализ баз данных ДНК для выявления сходства с известными организмами.
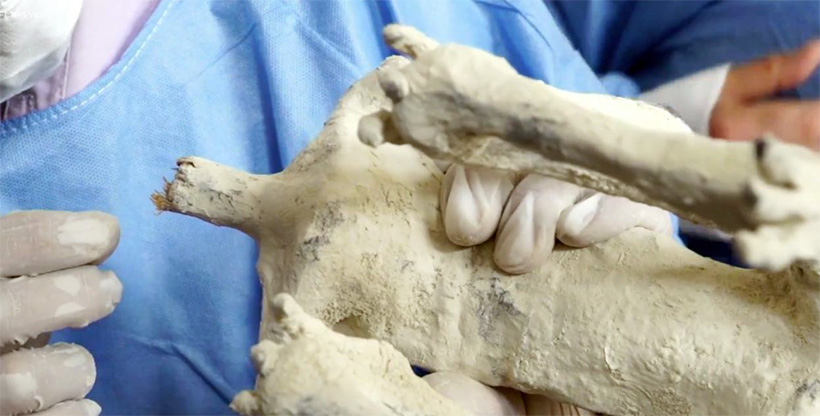
Рис.3.117. Извлечение образцов из шеи Виктории
Для выявления возможных типов организмов, присутствующих в образцах Ancient0004 и Ancient0002 (Виктория) был проведен геномный ДНК скетчинг DNA sketching (Ondov et al., 2016), в котором используется сопоставление групп коротких фрагментов, k-mers, с доступными базами данных. Был использован программный пакет BBTools software.
Были проверены следующие организмы:
- Bacteria.
- Virus.
- Plasmids.
- Phages.
- Fungi.
- Plastid.
- Diatoms.
- Human.
- Bos Taurus.
- H penzbergensis.
- PhaseolusVulgaris.
- Mix2: Label for the following genomes:
- Lotus japonicus chloroplast, complete genome.
- Canis lupus familiaris cOR9S3P olfactory receptor family 9 subfamily S pseudogene (cOR9S3P) on chromosome 25.
- Vigna radiata mitochondrion, complete genome.
- Millettia pinnata chloroplast, complete genome.
- Curvibacter lanceolatus ATCC 14669 F624DRAFT_scaffold00015.15, whole genome shotgun sequence.
- Asinibacterium sp. OR53 scaffold1, whole genome shotgun sequence.
- Bacillus firmus strain LK28 32, whole genome shotgun sequence.
- Bupleurum falcatum chloroplast, complete genome.
- Alicycliphilus sp. B1, whole genome shotgun sequence.
- Bacillus litoralis strain C44 Scaffold1, whole genome shotgun sequence.
- Chryseobacterium takakiae strain DSM 26898, whole genome shotgun sequence.
- Paenibacillus sp. FSL R5-0490.
- Bacillus halosaccharovorans strain DSM 25387 Scaffold3, whole genome shotgun sequence.
- Rhodospirillales bacterium URHD0017, whole genome shotgun sequence.
- Bacillus onubensis strain 10J4 10J4_trimmed_contig_26, whole genome shotgun sequence.
- Radyrhizobium sp. MOS004 mos004_12, whole genome shotgun sequence.
- Bacillus sp. UMB0899 ERR1203650.17957_1_62.8, whole genome shotgun sequence.
- Vertebrates: Label for the following genomes:
- Amblyraja-radiata_sAmbRad1_p1.fasta.
- bStrHab1_v1.p_Kakapo.fasta.
- bTaeGut1_v1.p_ZebraFinch.fasta.
- GCA_000978405.1_CapAeg_1.0_genomic_CapraAegagrus.fna.
- GCA_002863925.1_EquCab3.0_genomic_Horse.fna.
- GCF_000002275.2_Ornithorhynchus_anatinus_5.0.1_genomic.fna.
- GCF_000002285.3_CanFam3.1_genomic.fna.
- Macaco_GCF_000772875.2_Mmul_8.0.1_genomic.fna.
- rGopEvg1_p1_Gopherus_evgoodei_tortuga.fasta.
- Protozoa.
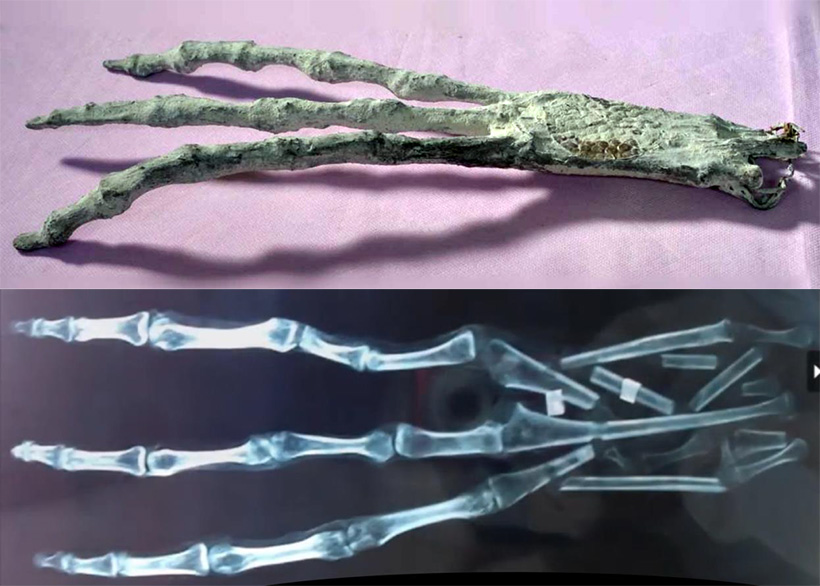
Рис.3.118. Изображение и рентгенограмма двух трехпалых рук
После всех фильтров было получено 27974521 ридов для Ancient0002 и 304785398 ридов для Ancient0004. Это показывает, что 27% ДНК из образца Ancient0002 и 90% ДНК из образца Ancient0004 не может быть идентифицировано с анализированными образцами ДНК организмов из доступных баз данных.
Следующий этап анализа был выполнен при помощи программ megahit v1.1.3 (Li et al., 2016). Был получен следующий результат:
- Ancient0002: 60852 contigs, total 50459431 bp, min 300 bp, max 24990 bp, avg 829 bp, N50 868 bp, 884,385 (5.39%) assembled reads.
- Ancient0003: 54273 contigs, total 52727201 bp, min 300 bp, max 35094 bp, avg 972 bp, N50 1200 bp, 20,247,568 (65.69%) assembled reads.
Результат анализа представлен на рисунке.
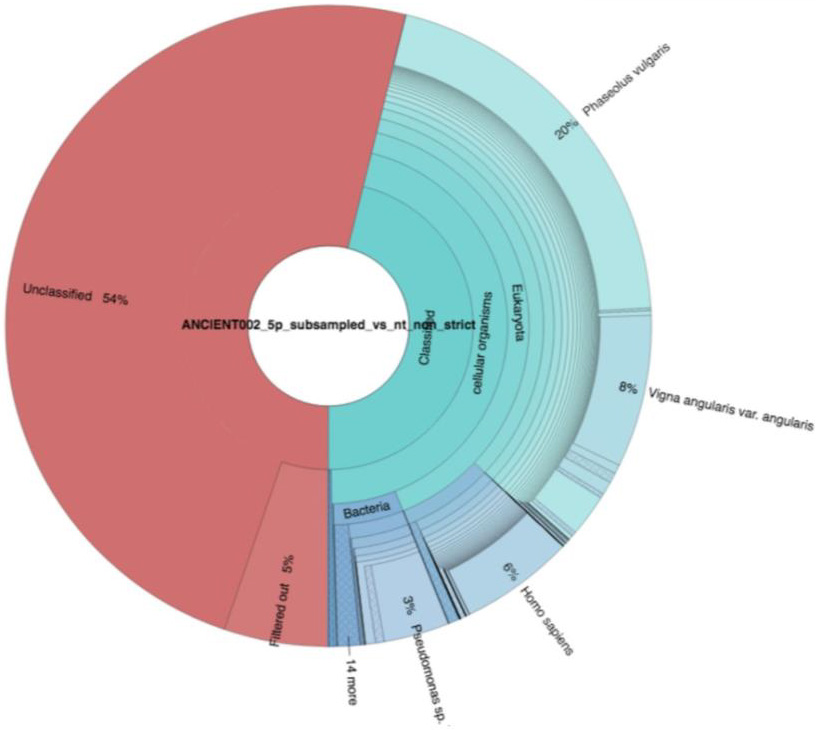
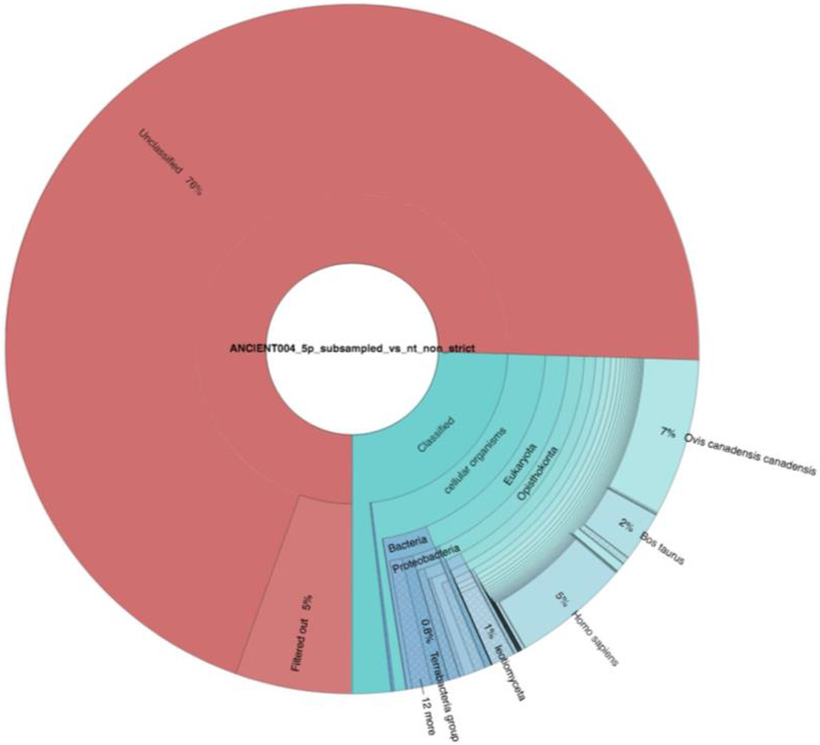
Рис.3.116. Соотношение классифицированных ридов для 28073655 ридов Ancient0002 (верхний граф) и 25084962 ридов Ancient0004 (нижний граф) в сравнении с базой 34904805 ДНК, представляющей 1109518
таксономических групп
Заключение
В результате проведенного анализа было показано отсутствие соответствия образцов Ancient0002 и Ancient0004 (Виктория) геному человека, в то время как образец Ancient0003 хорошо соответствует человеческому.
Комментарий Короткова К.Г.
Отметим, что трехпалая рука принадлежала большому существу, сопоставимому по размеру с Марией, и полученный результат соответствует результату ДНК анализу Марии. Виктория является представителем «маленьких существ», и полученный результат показывает, что их ДНК не соответствует никаким современным земным существам. Естественно, мы не имеем данных о древних существах, исчезнувших за миллионы лет.
Ссылки
- Corvelo, A., Clarke, W. E., Robine, N., & Zody, M. C. (2018). taxMaps: comprehensive and highly accurate taxonomic classification of short-read data in reasonable time. Genome Research, 28(5), 751–758.
- Gamba, C., Hanghøj, K., Gaunitz, C., Alfarhan, A. H., Alquraishi, S. A., Al-Rasheid, K. A. S., … Orlando, L. (2016). Comparing the performance of three ancient DNA extraction methods for high-throughput sequencing. Molecular Ecology Resources, 16(2), 459–469.
- Huang, W., Li, L., Myers, J. R., & Marth, G. T. (2012). ART: a next-generation sequencing read simulator. Bioinformatics , 28(4), 593–594.
- Li, D., Luo, R., Liu, C.-M., Leung, C.-M., Ting, H.-F., Sadakane, K., … Lam, T.-W. (2016). MEGAHIT v1.0: A fast and scalable metagenome assembler driven by advanced methodologies and community practices. Methods , 102, 3–11.
- Ondov, B. D., Treangen, T. J., Melsted, P., Mallonee, A. B., Bergman, N. H., Koren, S., & Phillippy, A. M. (2016). Mash: fast genome and metagenome distance estimation using MinHash. Genome Biology, 17(1), 132.
- Schubert, M., Ermini, L., Der Sarkissian, C., Jónsson, H., Ginolhac, A., Schaefer, R., … Orlando, L. (2014). Characterization of ancient and modern genomes by SNP detection and phylogenomic and metagenomic analysis using PALEOMIX. Nature Protocols, 9(5), 1056–1082.
- Weissensteiner, H., Forer, L., Fuchsberger, C., Schöpf, B., Kloss-Brandstätter, A., Specht, G., … Schönherr, S. (2016). mtDNA-Server: next-generation sequencing data analysis of human mitochondrial DNA in the cloud. Nucleic Acids Research, 44(W1), W64–W69.
- Zhang, J., Kobert, K., Flouri, T., & Stamatakis, A. (2014). PEAR: a fast and accurate Illumina Paired-End reAd mergeR. Bioinformatics , 30(5), 614–620.
Материалы предоставлены Константином Георгиевичем Коротковым (доктор технических наук, профессор, Университет Информационных Технологий, Механики и Оптики) и Дмитрием Владиславовичем Галецким (кандидат медицинских наук, Первый Санкт-Петербургский Государственный Медицинский Университет им. И.П. Павлова) специально для сайта «Мир тайн».
Другие статьи по теме:
- Результаты исследований ДНК тканей «Марии» и «Вавиты»
- Результаты исследований образцов тканей «Марии» и «Вавиты»
- Инопланетные мумии из Перу: хронология поисков и исследований
- Инопланетные мумии из Перу: объекты изучения
- Инопланетные мумии из Перу: конгресс
- Российские учёные подтвердили изучение тел внеземных существ
- Трёхпалая мумия пришельца найдена в Перу
- Krawix опубликовал новые находки из перуанской пустыни Атакама
- Трёхпалая рука показана общественности в Перу в январе 2017 года
- Мумифицированные останки пришельцев найдены в Перу в 2016 году









